
宏基因组测序技术已经广泛运用于微生物的研究当中,研究者使用计算方法从测序数据中重构出大量来自于人类,动物以及环境的微生物基因组用于后续的分析研究。通常的处理流程包括测序数据的组装(Assembly)和分箱(Binning)。目前广泛使用的宏基因组分箱方法均为无监督(不依赖于参考基因组)的方法,忽略了参考基因组中的信息。复旦大学类脑研究院生物医学AI团队提出了一个半监督的宏基因组分箱算法.SemiBin(https://github.com/BigDataBiology/SemiBin),使用深度孪生网络结合了参考基因组中的信息,在GMGC(Global Microbial Gene Catalog) 数据库的10个环境中,均超越了目前最好的分箱算法。
SemiBin在多个模拟数据和真实数据集上取得了良好性能。在人类肠道、狗肠道、海洋以及土壤的真实环境中,使用单样本分箱情况下,相比于目前最好的分箱工具,SemiBin能够多重构出41.2%,72.0%,48.7%和61.0%的高质量基因组;使用多样本分箱,SemiBin能够多重构出17.5%,11.0%,30.7%和171.4%的高质量基因组(见图1)。SemiBin已经能够得到很好的分箱结果,但是物种注释以及模型的训练仍然需要很大的计算资源,限制了SemiBin在大型数据分析中的应用。为此,提出了SemiBin(pretrain),从多个样本中进行预训练,然后将训练得到的模型直接应用于其他的样本,可以大大加快分箱的速度。与此同时,SemiBin(pretrain)可以得到更好的结果,相比于Metabat2,SemiBin(pretrain)可以多重构出60.4%,99.2%,48.0%和74.6%的高质量基因组。
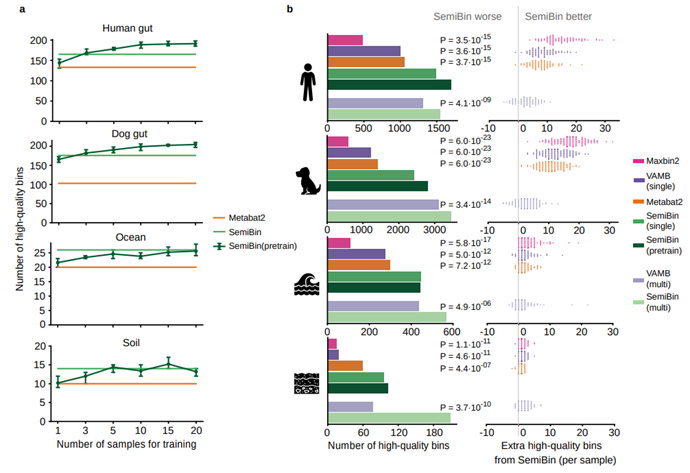
图1. SemiBin在真实数据上的结果。(a)不同数量的样本用于预训练对于结果的影响;(b)4个真实环境中的benchmark结果。
这一研究成果已于2022年4月28日发表在Nature Communications期刊。复旦大学类脑智能科学与技术研究院生物医学AI团队的博士生潘少军是本研究的第一作者,赵兴明教授和Luis Pedro Coelho青年研究员为本论文的共同通讯作者。