
2023年4月27日, 实验室团队在《生物信息学简讯》(Briefings in Bioinformatics)杂志在线发表了题为《基因组解析肠道宏基因组学的计算策略研究》(“A survey on computational strategies for genome-resolved gut metagenomics”)的文章。
恢复高质量的宏基因组组装基因组(HQ-MAGs, high-quality metagenome-assembled genomes)对于探索微生物组成和微生物与表型的关联至关重要。然而, mNGS、 PacBio、Nanopore、 metaHiC等多种测序平台和配套的计算工具提供了数十种恢复HQ-MAGs的策略,使得研究人员难以选择,因此需要进行广泛的评估。
在本工作中,研究团队系统地评估了40种流行的计算工具和测序平台的组合(即策略),涉及8种宏基因组组装工具、8种分箱工具和4种测序技术,包括短读段、长读段和metaHiC测序。根据测序数据的可用性,团队确定了用于单个任务(例如宏基因组组装和分箱)和组合(例如生成更多HQ-MAGs)的最佳工具。研究发现混合组装与基于metaHiC的分箱的组合表现最好,其次是混合组装和长读段组装。更重要的是,长读段测序和metaHiC测序都将更多的可移动元件和抗生素抗性基因与细菌宿主联系起来,并提高了公共人类肠道参考基因组的质量,其中32%(34/105)的HQ-MAGs质量比统一人类胃肠道基因组目录第2版(UHGG2)中的质量更好或者是新的HQ-MAGs。
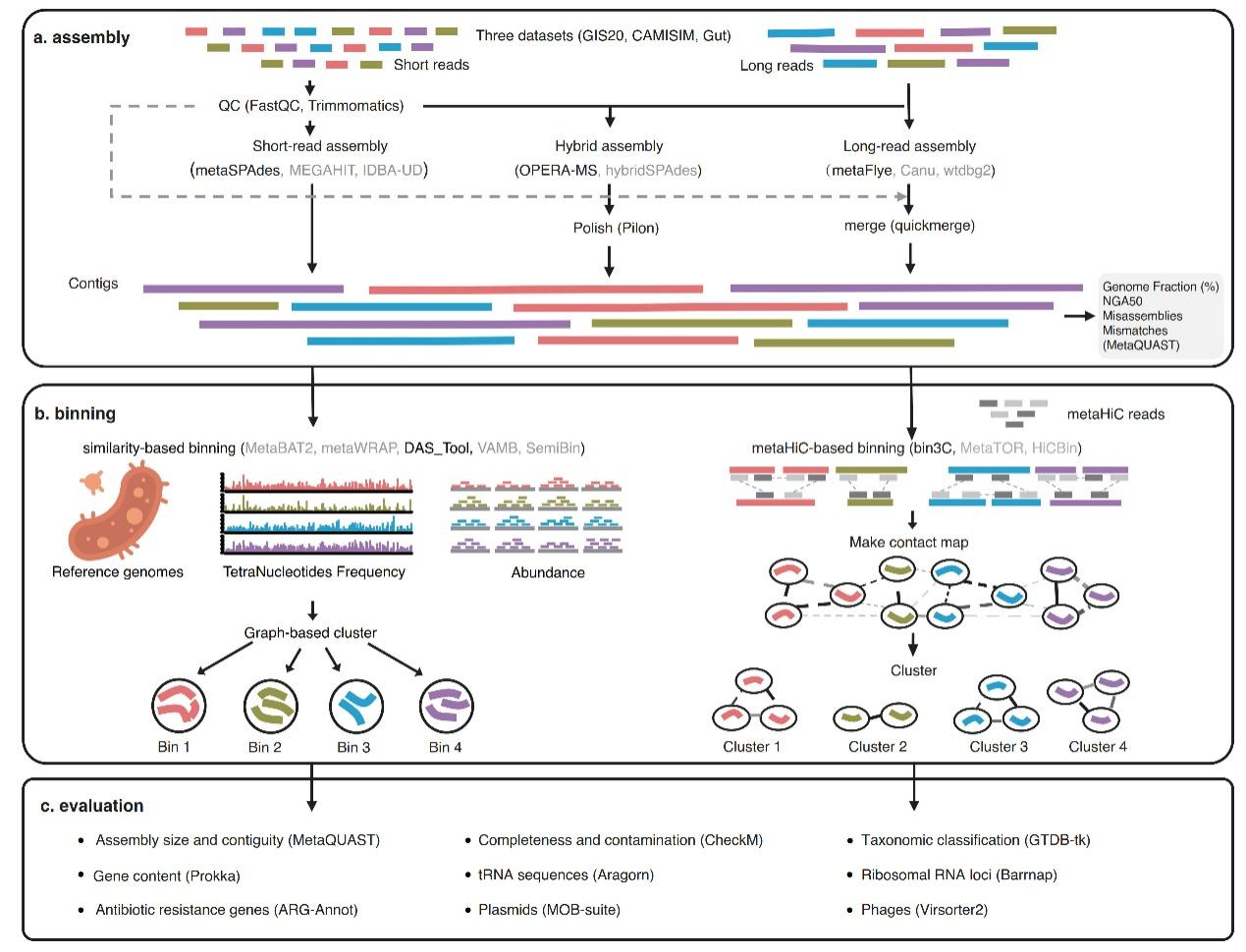
图1.使用短读段、长读段和metaHiC测序数据对基因组解析宏基因组学策略进行系统评估的流程图。
复旦大学生物医学AI实验室的博士生贾龙豪是本研究的第一作者,赵兴明教授和华中科技大学的陈卫华教授为本论文的共同通讯作者。生物医学AI实验室的博士生董彦祺和华中科技大学的博士生吴英健和陈景超也为了本研究做出了重要贡献。
原文链接:
https://academic.oup.com/bib/advance-article/doi/10.1093/bib/bbad162/7145904